UCSC Xena主要解决两个问题:
- genomics数据的可视化,无论是大型公开数据库还是自己的私人数据。
主要数据来自TCGA、ICGC、CCLE等;
所有可以进行可视化的数据都可以下载,而且这些数据被预处理过,可以直接下载使用;
可以比较方便的可视化私人数据。
这里对于私人数据集,基于可利用资源、安全性的诸多考虑,Xena将此分析过程放在本地进行。所以其交互模式如下图所示:
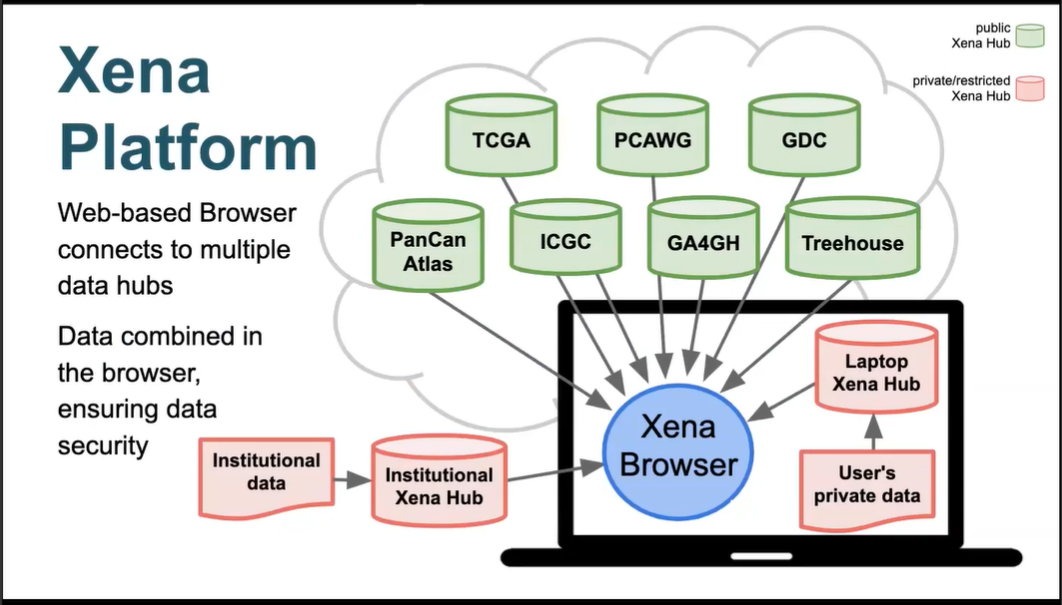
- 将私人数据和公开数据进行合并分析。
- 可以查看的数据类型有:Copy number variation、DNA methylation、Gene and exon expression、Protein expression、miRNA expression、Positional mutation和Phenotype/annotations;
- 另外一个整合的web分析工具--MuPIT,可以在3D蛋白质空间中查看变异。
UCSC Xena总体来说,为geneomics研究提供了一个快速实现的平台。一些简单的、即时性的可视化内容,或者分析单个或几个gene,可以直接使用UCSC Xena实现,无需下载、预处理数据。
当然,其功能相对于使用R、python分析来说,还是比较简单的,并且其主要目的在于快速的可视化,而不是复杂的数据分析。所以对于稍微复杂的数据分析流程、成百上千个genes的可视化等任务,其可能无法完成。而且在中国大陆使用的话,可能需要找个合适的时间(比如上午)。
UCSC Xena的各种功能众多,并且还储存了大量的可用precompiled data,所以这里我会分多篇文章进行介绍:
- ,UCSC Xena的基本操作等也在此处进行了介绍
- UCSC 统计图示,这个比较简单,就直接链接到tutorial了
另外,UCSC Xena的一些重要的内容,我也以链接的形式放到下面,方便访问:
UCSC Xena介绍的其他的项目:
- TumorMap,由UCSC开发的另一个项目。
- MuPIT,可以可视化mutation所带来的蛋白质的影响。
- Transcript View,可视化肿瘤样本(TCGA)和正常样本(GTEx)间的差异。
- Gene Sets Tool,比较了两个队列间癌症相关基因的somatic mutation和copy number。



