Predicting the sequence specificities of DNA- and RNA-binding proteins by deep learning
- 杂志: Nature Biotechnology
- IF:
- 分区:
Introduction
- DNA,比如控制转录或可变剪切,而其功能和其特异性的序列相关。
- position weight matrices(PWMs)可以用来描述这种序列特异性,并且易于解释。
- 一些新的、更加复杂的技术能够更加准确的来描绘序列特异性,本研究提出了一种基于Deep Learning的技术,称为DeepBind,能够发现新的patterns。
使用高通量数据来进行序列特异性的建模有下面几个问题:
- 来自不同技术的数据有着不同的形式。
- 高通量数据一般比较多,有10000-100000条序列。
- 不同类别的数据有着不同的偏移和限制。
而DeepBind依次解决了上述问题:
- 其可以应用于微阵列数据或序列数据
- 使用GPU来加快训练
- 在这个数据间泛化良好,甚至没有进行校正
- 可以允许一定的噪声
- 可以自动化的进行训练,减少了手动调参的工作
- 最后,可以类似PWMs一样进行可视化,从而提供一定的可解释性
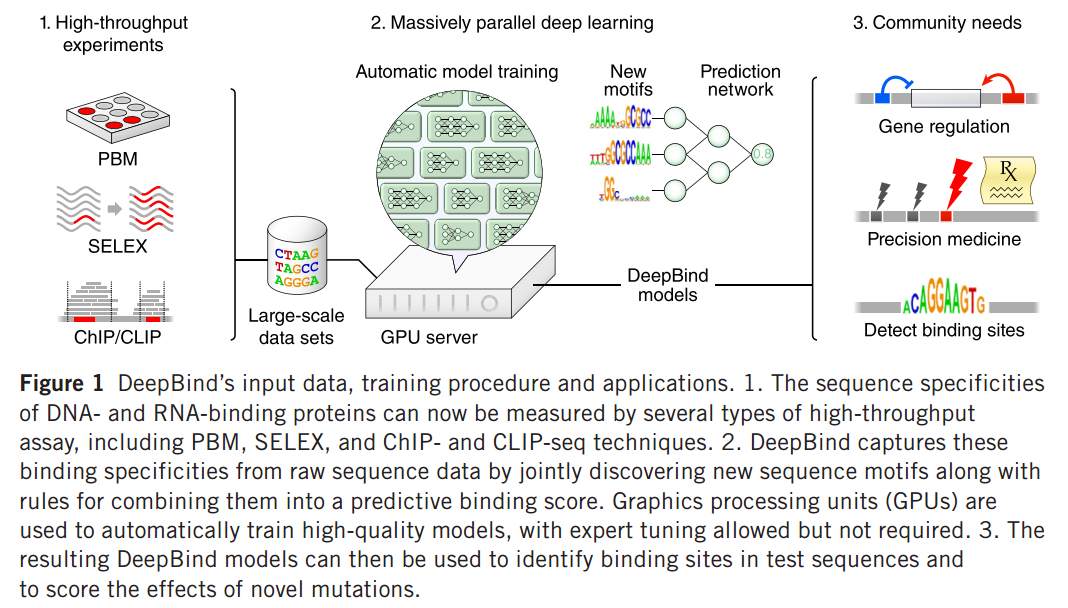
Methods
数据
序列长度14-101nt不等,每条序列有一个binding score,可以是real-value或binary class labels。
模型
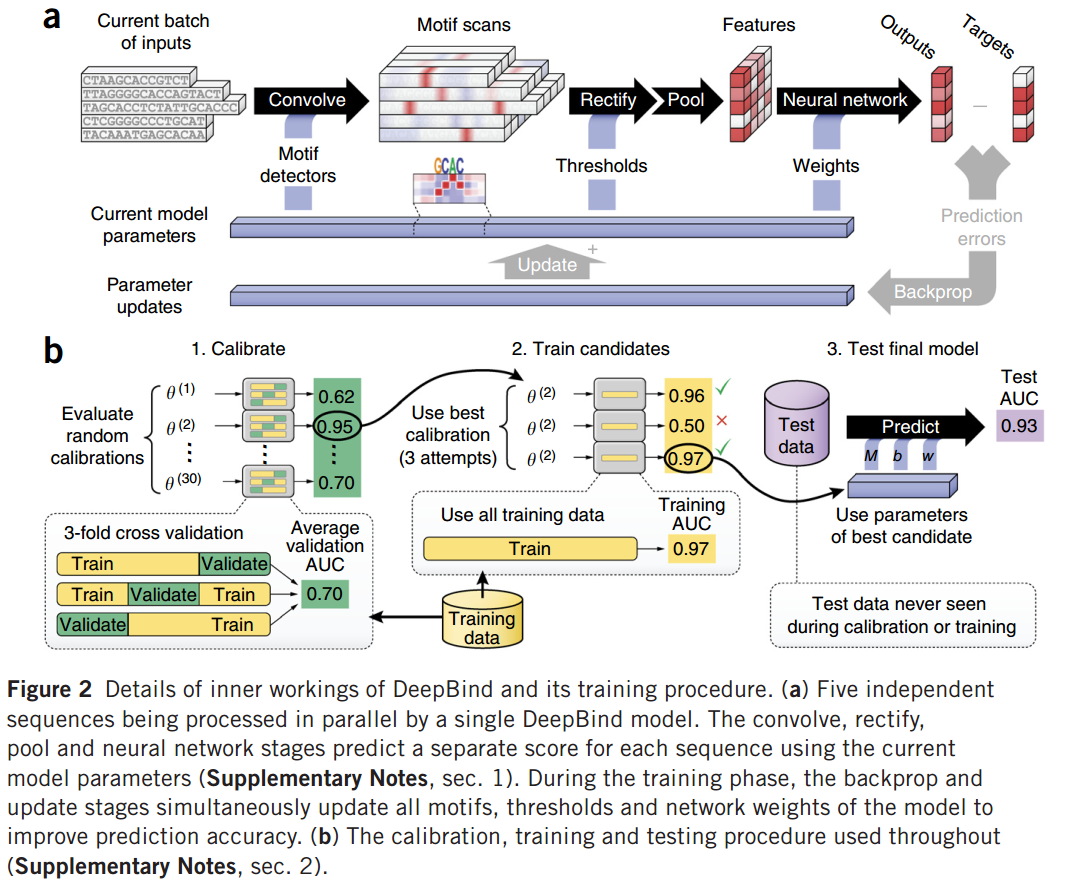
首先经过一层CNN,然后进行global max pooling,这样每个序列得到一个相同长度的表示向量。
将此向量送入MLP进行预测,然后和binding score计算loss,反向传播进行训练。loss为:

为了能够进行自动化地训练,这里:
- 对于每个需要训练的模型,随机采样30组参数;
- 3-CV训练并计算测试集误差,选择最好的那组参数。
完整的参数列表在supplementary notes中
整个模型的训练使用了12 terabases的序列数据,源代码,其中包括了927个DeepBind模型,对应538个确定的转录因子和194个RBPs。
评价
DNA模型使用revised DREAM5 TF-DNA Motif Recognition Challenge的PBM数据进行验证。



